NextDenovo(https://github.com/Nextomics/NextDenovo)
由希望组研发总监+联合创始人胡江带领的团队历时2年研发的一款集比对、矫正、组装于一体的基因组组装软件,针对三代测序(Nanopore、PacBio)数据进行高效矫正和精确组装,极大减少计算资源和运行时间,同时组装出高质量基因组。
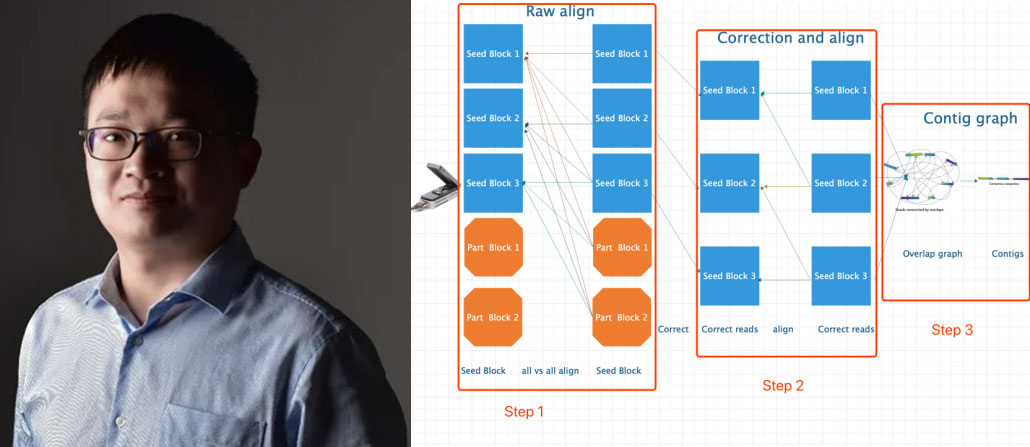
图1 NextDenovo 作者和NextDenovo flowchart
NextDenovo 一经开源引起国内外广泛使用和报道。截止2020年5月NextDenovo Github(https://github.com/Nextomics/NextDenovo)下载次数超过2000次。
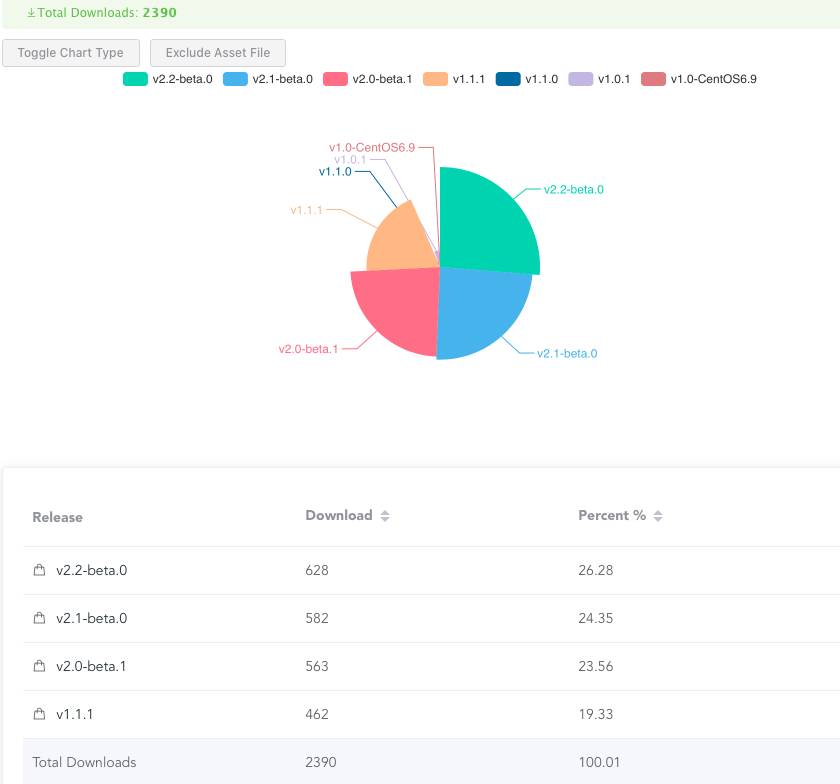
图2 Github 上NextDenovo版本迭代以及下载次数统计

图3 国内外媒体对NextDenovo 报道
对于复杂基因组(高杂合植物Plant1、高同源多倍体Plant2、超大基因组Plant3/Plant4)NextDenovo表现非常好的性能。
表1 NextDenovo对于复杂基因组组装表现
| Species | Genome size(Gb) | Contig N50(Mb) | Contig number(#) | Total length(bp) |
| Plant1 | 2.3 | 66.3 | 275 | 2,037,039,384 |
| Plant2 | 0.77 | 59.7 | 185 | 780,786,253 |
| Plant3 | 11.02 | 5.02 | 3,641 | 10,417,871,370 |
| Plant4 | 10.76 | 93.26 | 329 | 10,759,349,041 |
截止2020年5月使用NextDenvo发表文章多篇,包括Nature Plant、GigaSciecne等知名期刊。
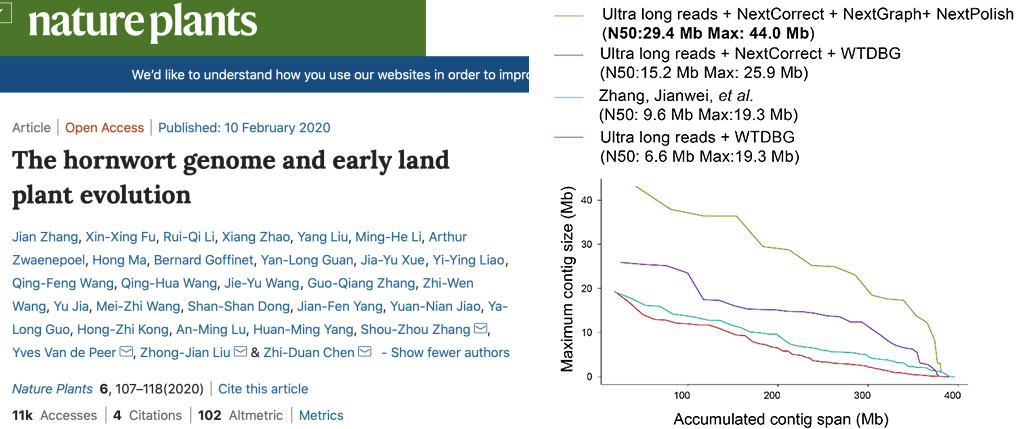
图4 使用NextDenovo 发表文章信息展示
NextPolish(https://github.com/Nextomics/NextPolish)
由希望组研发团队开发的Next系列软件中针对长读长(Nanopore、Pacbio)组装软件(NextDenovo、Canu、Miniasm等)组装得到的基因组进一步单碱基polish小工具,它可高效矫正三代下机数据组装得到基因组的单碱基错误,进一步提高单碱基准确性。
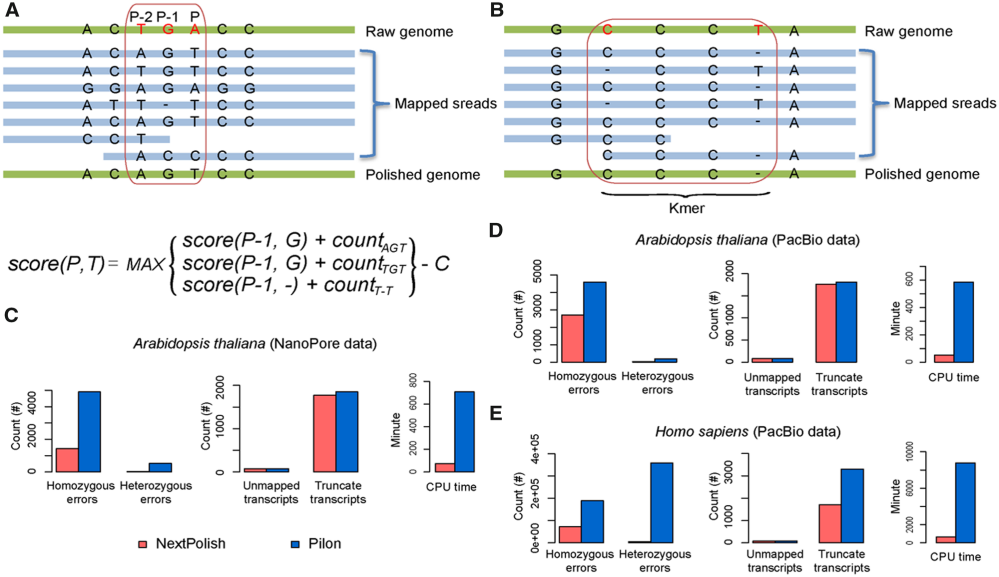
图 1 NextPolish算法原理以及性能比较
该工具采用K-mer得分链和K-mer计数算法,在运行速度、校正精度及消耗资源等方面均优于同类软件(表1),NextPolish目前已在Bioinformatics期刊正式发表。
| Species | Platform | Software | Polishing rounds (#) | Total length (bp) | Misassemblies | Mismatches per 100 kbp | Indels per 100 kbp | Total mismatches | Total indels | CPU time used for polishing (minutes) | CPU time used for alignmenta (minutes) | Maximum RAM (GB) |
| Homo sapiens (Chr.1) | SMRT | – | 0 | 224,780,032 | 6 | 17.82 | 121.45 | 38,286 | 261,011 | – | – | – |
| NextPolish | 1 | 224,716,364 | 6 | 2.38 | 0.81 | 5,107 | 1,736 | 21 | 342 x 4 (rounds) | 7.21 | ||
| 2 | 224,716,261 | 6 | 2.26 | 0.71 | 4,863 | 1,527 | ||||||
| Pilon | 1 | 224,715,544 | 6 | 2.92 | 1.6 | 6,275 | 3,447 | 484 | 46.96 | |||
| 2 | 224,715,452 | 6 | 2.39 | 1.36 | 5,134 | 2,917 | ||||||
| 3 | 224,715,529 | 6 | 2.31 | 1.28 | 4,974 | 2,754 | ||||||
| 4 | 224,715,464 | 6 | 2.31 | 1.25 | 4,957 | 2,684 | ||||||
| Racon | 1 | 224,702,036 | 9 | 3.08 | 1.97 | 6,625 | 4,242 | 1,122 | 101 x 4(rounds) | 83.02 | ||
| 2 | 224,691,554 | 9 | 2.91 | 1.16 | 6,249 | 2,494 | ||||||
| 3 | 224,685,532 | 11 | 2.87 | 1.08 | 6,177 | 2,312 | ||||||
| 4 | 224,681,897 | 11 | 2.64 | 1 | 5,684 | 2,148 |
表2 NextPolish、Pilon和Racon的性能比较
- BWA MEM was used to do alignment for NextPolish and Pilon, while Racon used Minimap2 by default.
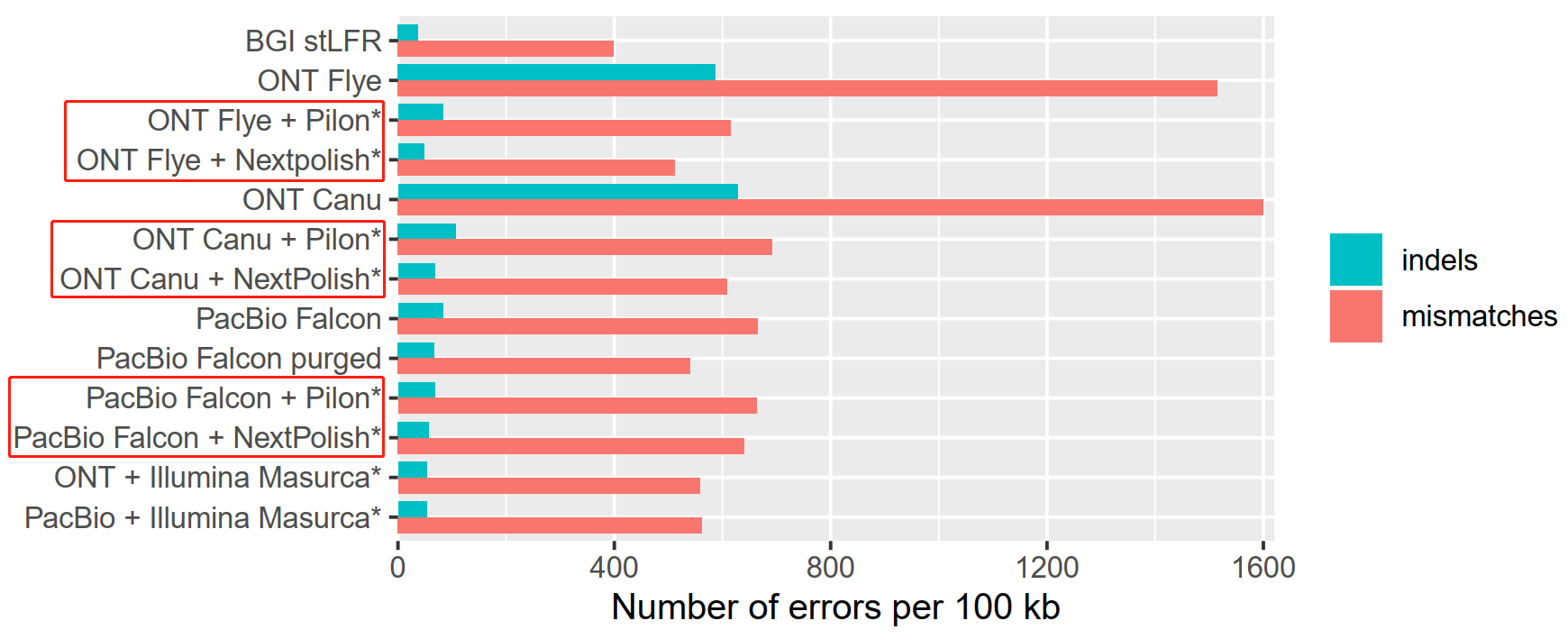
图2 三代polish矫正软件矫正结果比较
[1] Jiang H , Junpeng F , Zongyi S , et al. NextPolish: a fast and efficient genome polishing tool for long-read assembly[J]. Bioinformatics(7):7.



