产品优势
相比其他短读长测序技术
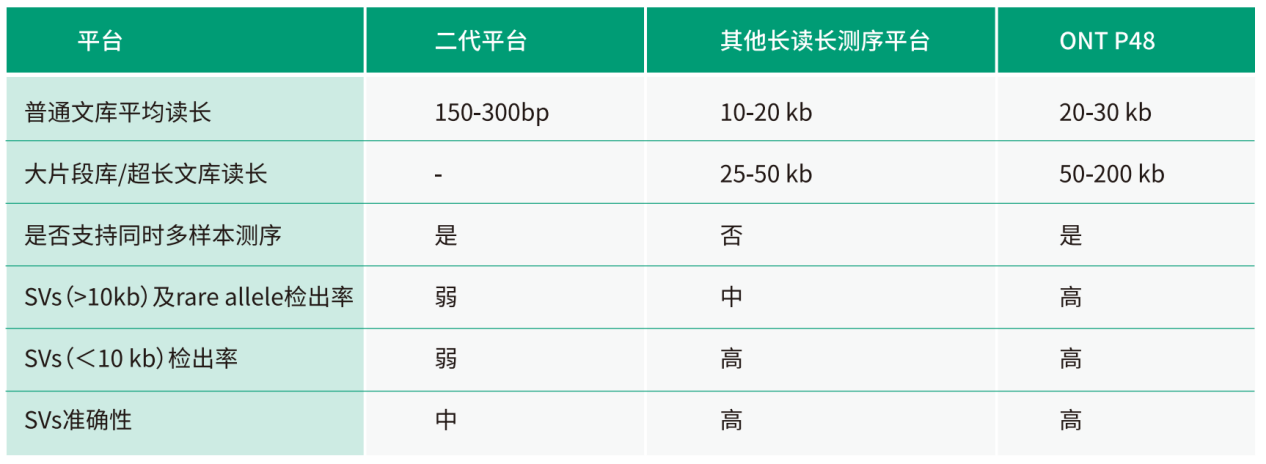
1.读长长,最长reads可达2Mb,可轻松跨越大部分重复序列区或杂合区域。
2.建库和测序过程中均无PCR过程,无GC偏好性,对基因组覆盖更完整及更均一。
3.能够检测较大的 insertion,且可以给出insertion的序列信息,并对insertion序列进行序列类型的鉴定看其是否是由于转座子等重复序列插入引起。
4.能够检测其他更复杂结构变异 ,且鉴定准确率更高。
相比其他长读长测序技术
1.reads长度更长,检测敏感性更高。
其他长读长测序平台需要多个cell才能达到SVs calling所需要的数据量,无法满足群体基因组同时测序多个动植物基因组样本的要求。而 ONT PromethlON48(P48)在群体基因组研究上存在明显的周期优势,P48能最多同时并行多达48个样本,且单Cell产量能达到大部分物种30×的数据量需求,极大压缩了群体测序所需时间,在群体测序周期方面有明显优势。
2、测序通量更高,节省研究周期
ONTP48的平均read N50长度在25-40kb,而Ultra-long reads的建库方法其reads N50长度甚至可达100kb以上,从而使得ONTP48在重复区域有更高的检测能力,能够覆盖更多的完整SVs,提升对大片段SVs(>10kb)和稀有SVs检测的敏感性。
方案策略

分析内容
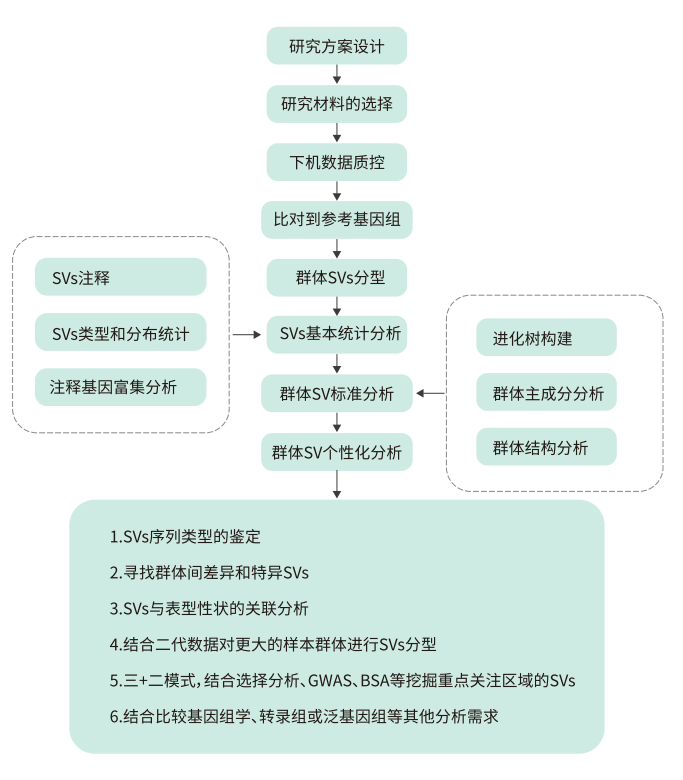
结果展示
结构变异检测
在比对到参考基因组序列的基础上,通过结构变异检测分析软件检测每个样本全基因组范围内所有潜在的SV变异。
图1 SV染色体分布图
重要SV相关的基因富集分析
发生在基因exon以及上下游调控区的SV,对基因更有可能造成较大的影响。针对注释结果中发生在exon和上下游区域的SV相关的基因进行富集分析。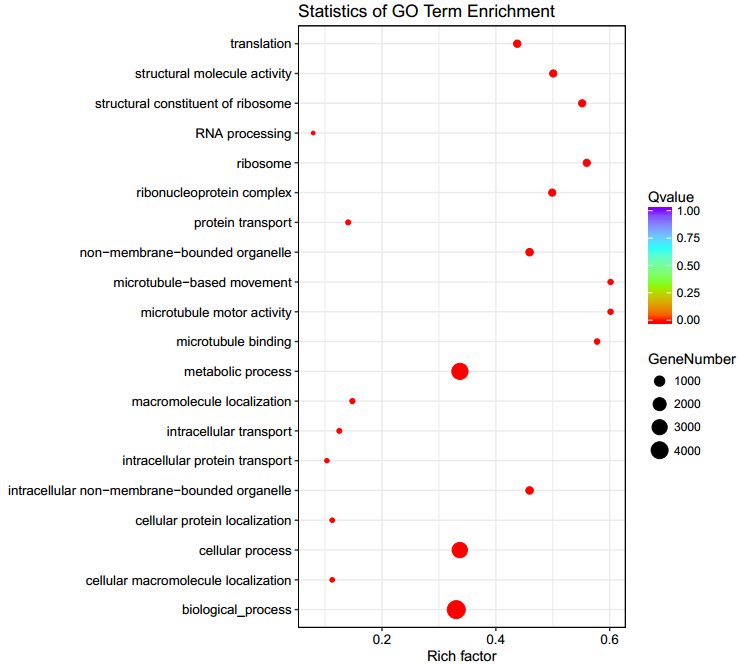
图2 GO分析统计。横坐标为Rich Factor,表示富集上的基因占注释到的基因的百分比;纵坐标表示富集上的条目;点的大小表示富集上的基因个数,颜色表示的Qvalue值,越低越显著。
群体结构分析
群体结构(population structure),群体结构是群体间差异的直观体现,群体间差异越大,所展示出的结构越明显。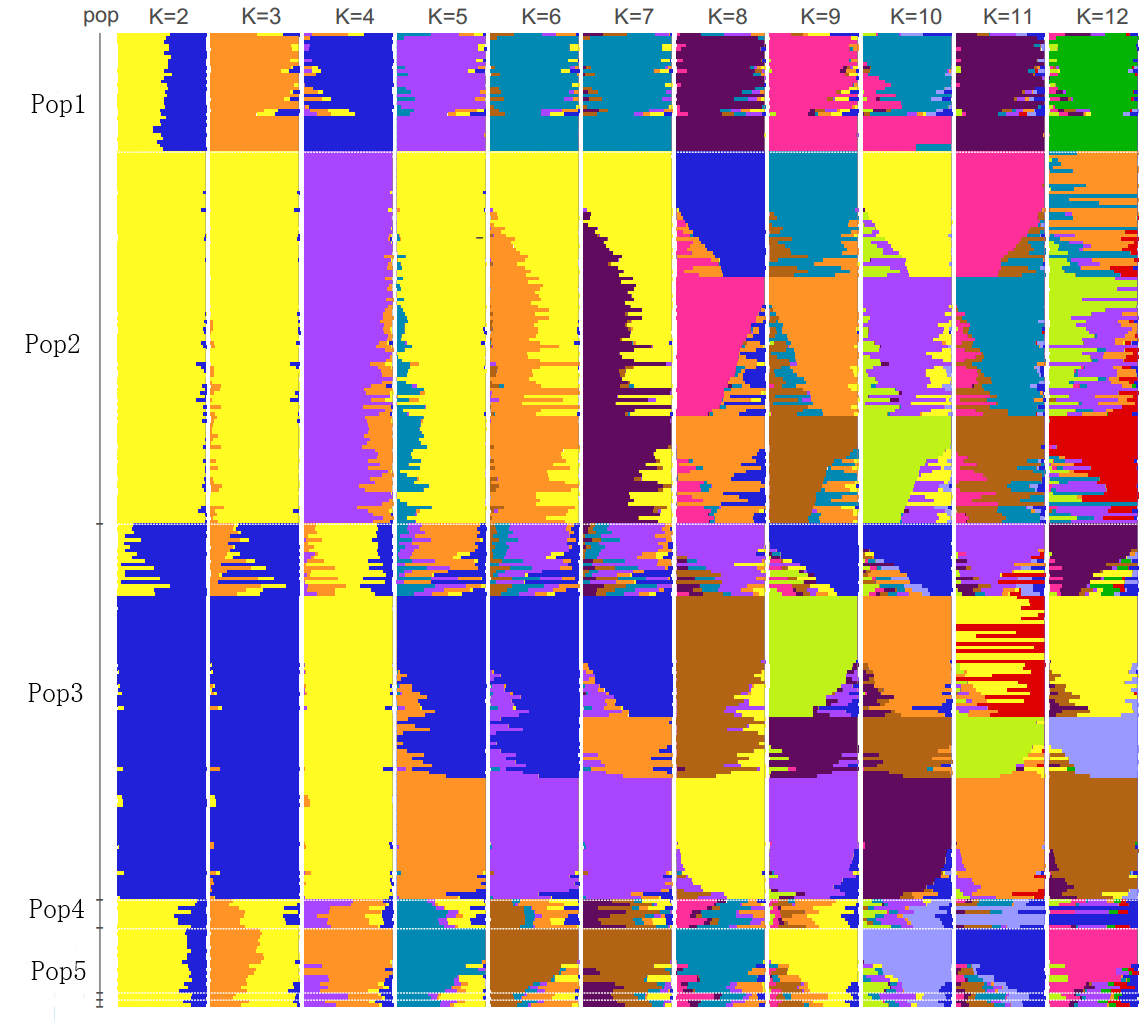
图3 群体结构整合图。竖着的一个子图是每个具体的K下的分群结果;横着是每个样本,样本在每个K中一一对应;颜色标注代表每个K下的一个群体信息,但K与K之间的颜色不具有任何关联关系,即K=4中的黄色与K=5中的黄色不代表同一个群体,没有任何关系。
群体间差异SV鉴定
Fst (Fixation index, 固定指数) 是衡量因遗传结构差异而引起群体的群体间分化(population differentiation)程度的常用指标。Fst的计算需要有至少两个群体,Fst值(1≥Fst≥0)越大表示群体间分化越大。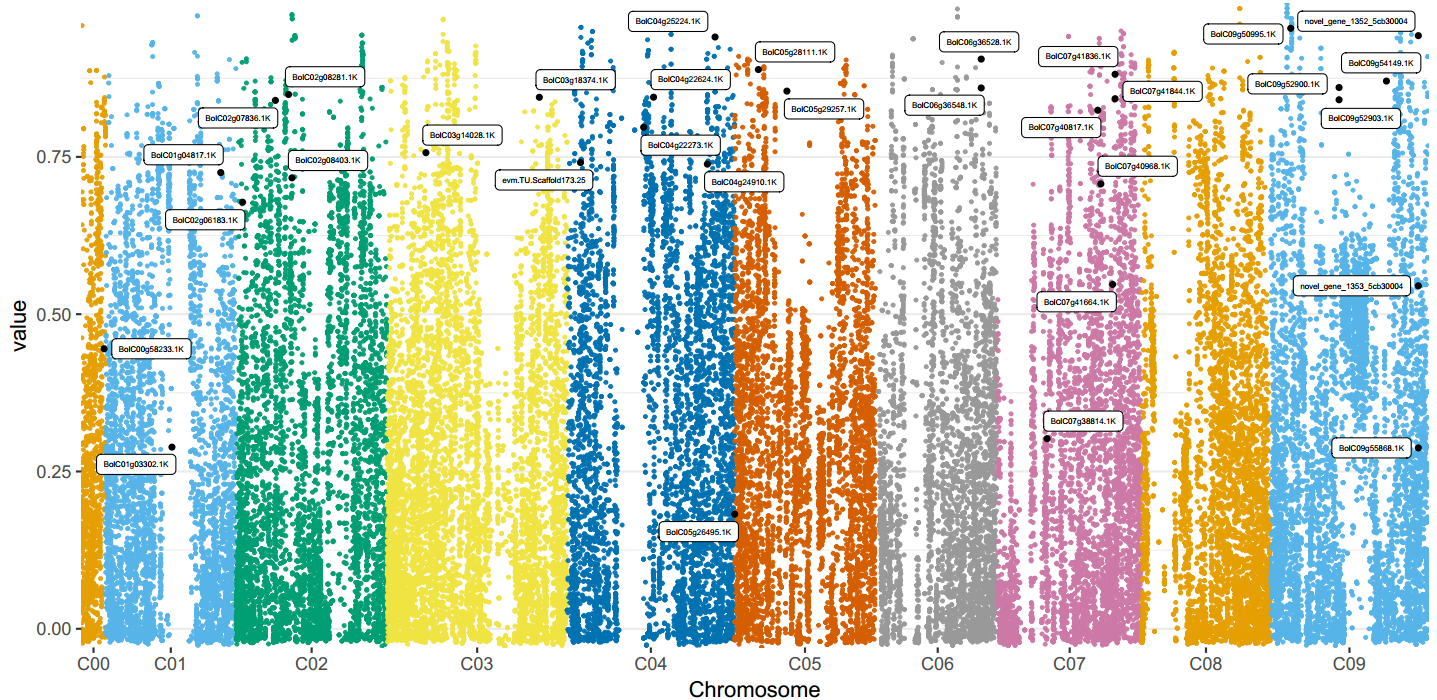
图4 Fst结合重点关注基因。图中每种颜色代表一条染色体的结果,每个点代表一个SV的Fst结果值;黑色点的位置为重点关注的SV所对应的Fst值,其旁边的基因为该SV注释到的基因。
案例解析
案例分析
案例分析
Nanopore测序识别人类基因组(Yoruban NA19240)中的结构变异
人类基因组中的结构变异(Structural variants SVs)定义为拷贝数或位置变化大于50bp的DNA区域,包括拷贝数变异(CNV)、倒位(Inversion)、易位(Translocation)、移动元件插入(Mobile element insertion)、重复序列扩增(Expansion of repetitive sequence)等。短读长技术对大多数SVs的检测效果很差,长读长测序技术的发展使准确检测并扩展基因组SVs成为可能。研究者利用Nanopore PromethlON测序平台,对人类Yoruban NA19240基因组进行了测序,利用这些数据对多个SVs比对、检测工具(LAST、Ngmlr、Minimap2、Sniffles、NanoSV等)进行了准确性和敏感性的测试评估,并讨论了以上工具的最佳参数,同时提供了高置信度和敏感性的SVs集合。其中比对工具Minimap2和结构变异检测工具Sniffles在该研究中表现出最高的准确性和计算效率。最后,该研究所描述的利用Nanopore PromethlON测序平台进行SVs检测的工作流程,可拓展至其他个人或群体进行长读长测序和SVs检测。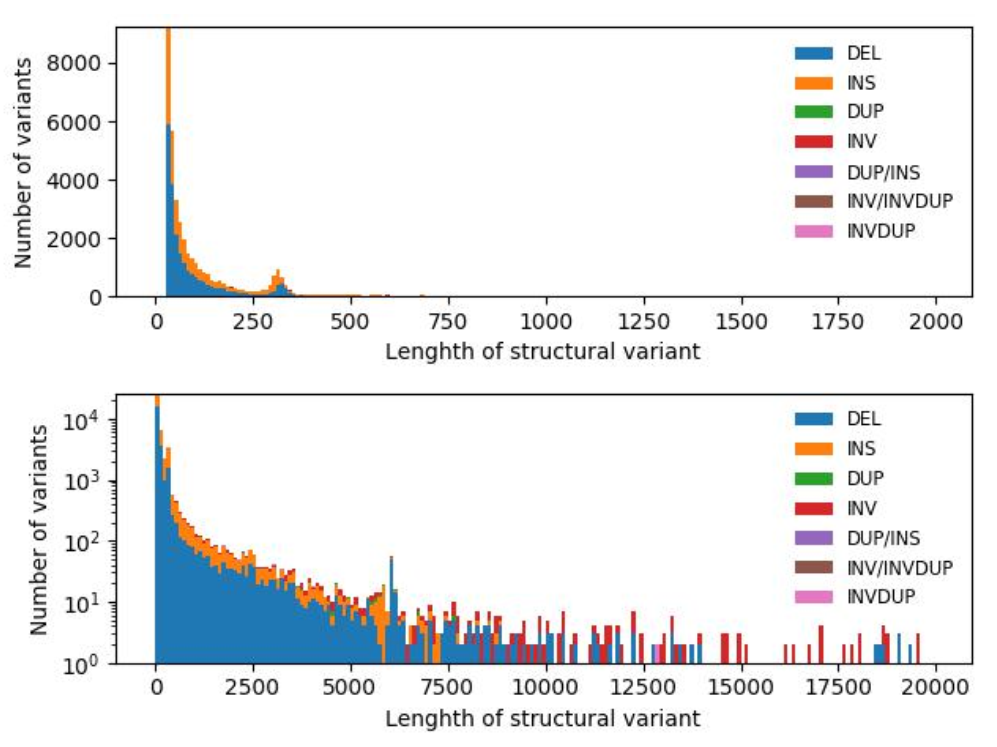
图1 不同类型SVs长度分布
葡萄驯化中结构变异的种群遗传学
目前植物研究中对SV的类型、大小、在个体中的分布及其种群动态所知甚少。了解这些动态对于了解SV对表型的贡献在GWAS关联分析中至关重要。本研究通过调查野生和驯化葡萄中群体水平SVs的变异情况来填补我们对植物基因组进化认知的空白。研究者首先利用三代测序技术组装了高杂合的“霞多丽”(Chardonnay)葡萄基因组,然后将该基因组与“赤霞珠”(Cabernet Sauvignon)葡萄基因组进行综合比较,利用基因组之间的SVs,对另外50个葡萄品种和19个野生祖先群体进行了SVs的分型,经过严格过滤后获得了481,096个SVs。利用该群体SVs集合,推断了不同类型变异的选择强度,其中TRAS和INVs的选择性更强,同时探讨了在栽培葡萄上由杂交向无性繁殖转变的效应。栽培葡萄与其野生祖先之间SV差异特别显著的基因区域,发现存在一个4.82Mb的倒位(b),这一变异与MybA基因的半缺位性共同导致具有白色浆果的微多丽葡萄的形成。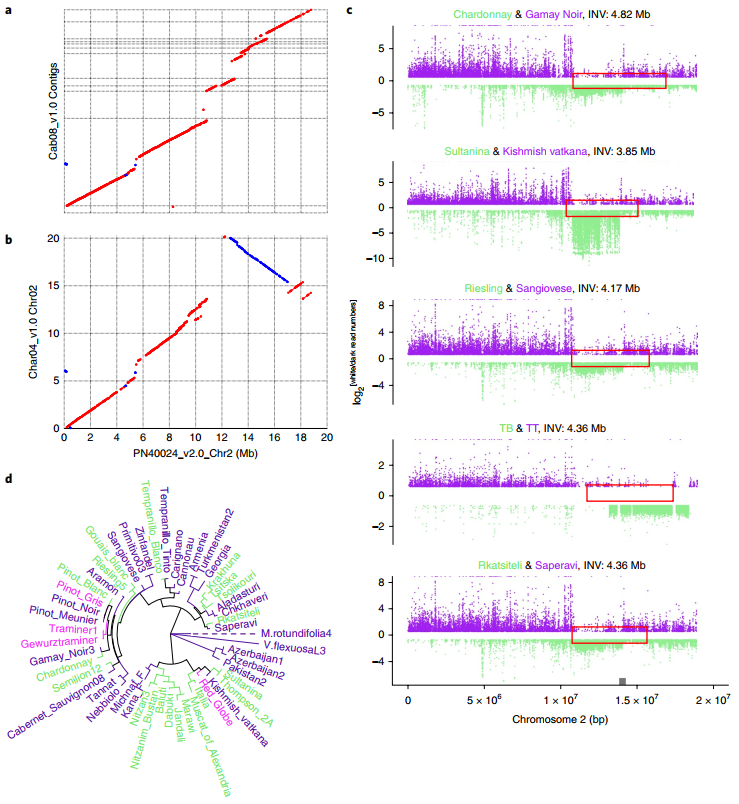
图2 栽培葡萄与其野生祖先之间4.82Mb倒位基因区域



