
近日,北京希望组与Sentieon联合宣布,双方决定在基因组数据分析的多个领域进行战略合作,持续推动包括二代和三代测序在临床诊断的产品落地。
双方合作的重点包括了以下四点:
1.联合推出基于PacBio HiFi数据的基因组重测序分析流程,变异检测范围包括了SNP,Indel和SV(结构变异);2.面向ONT最新推出的Q20+最新试剂数据,双方将进一步联合开发全面覆盖SNV/Indel/SV的变异检测产品;3.验证并部署基于Sentieon已经完成的MGI数据全基因组(WGS)分析流程;
4.将双方的合作纳入“希望诊断计划”,为该计划开发并搭建全方位的长、短读长结合的大数据变异分析平台。
GrandOmics长读长数据SV检测软件——GrandSV高质量的结构变异检测,是基因组医学的基石,无论是针对罕见病、肿瘤,还是辅助生殖,各种基因组医学的应用,均急切的需要新型的工具解决复杂的基因组结构变异检测问题。
长读长测序对于大片段的结构变异检测具有天然的优势。综合长读长数据分析算法中的序列拼接和序列比对算法,希望组开发了针对于长读长测序数据(PacBio HiFi和ONT)的SV检测软件——GrandSV。和以往长读长数据SV检测软件相比,兼顾了两种特征鲜明、截然不同的长读长数据(PacBio HiFi/ONT),具有更高的准确度和灵敏度。
利用准确度在99%左右的PacBio HiFi模拟数据来评估GrandSV与同类型软件cuteSV v1.0.9,Pbsv v2.4.0,Sniffles v1.0.12在人类基因组上鉴定结构变异(SV)以及变异分型(Genotyping)的准确性和灵敏度。图1的结果显示,GrandSV在5-30X的不同深度上效果都是最好的之一。此外,模拟Super Accuracy basecalling模式下的ONT数据分析结果也同样显示出了GrandSV的不俗表现。
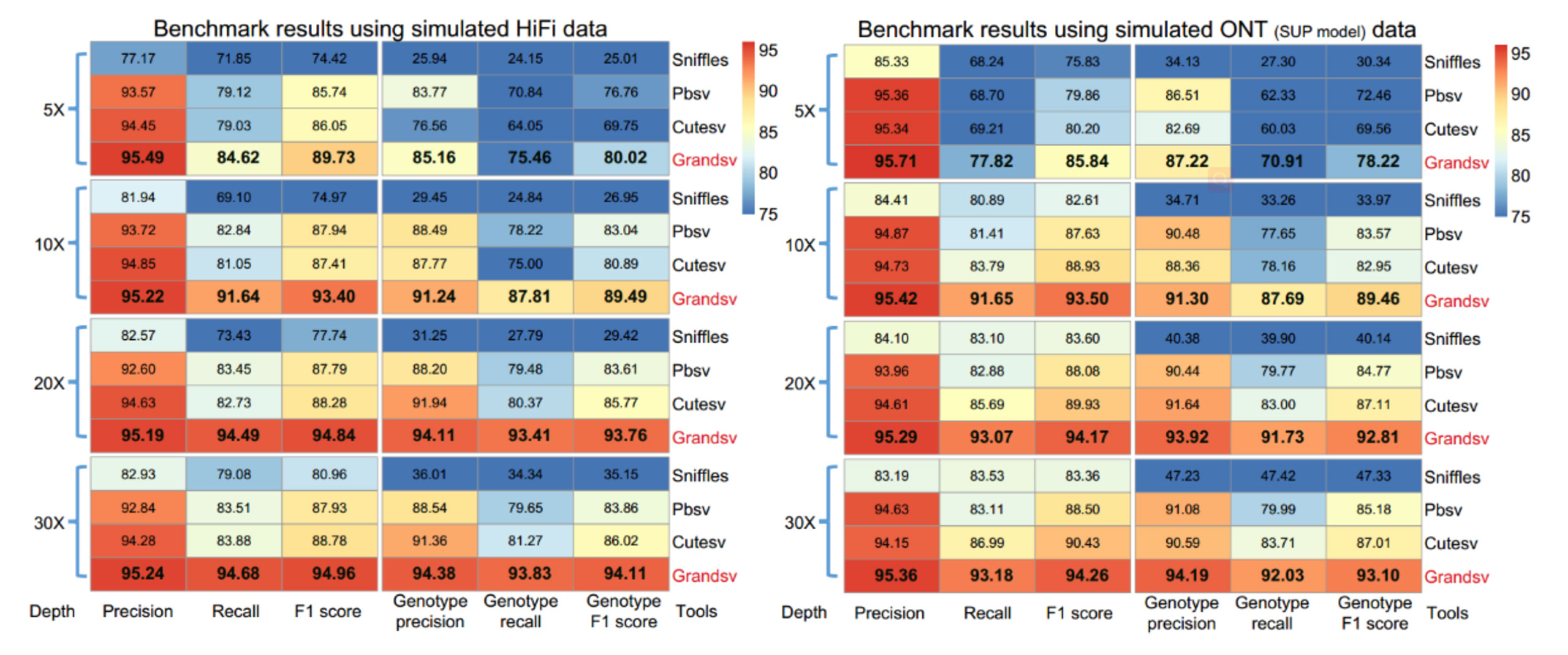
图1. Benchmark with simulated PacBio HiFi and ONT (SUP model) data
除模拟数据外,基于HG002的PacBio HiFi和ONT真实数据,并以Genome in a Bottle Consortium (GIAB) 团队发表的34,830个高置信区间当做真实的背景数据集来评估GrandSV表现。结果依然显示GrandSV的整体效果在5-30X的不同深度上都是最优的之一。
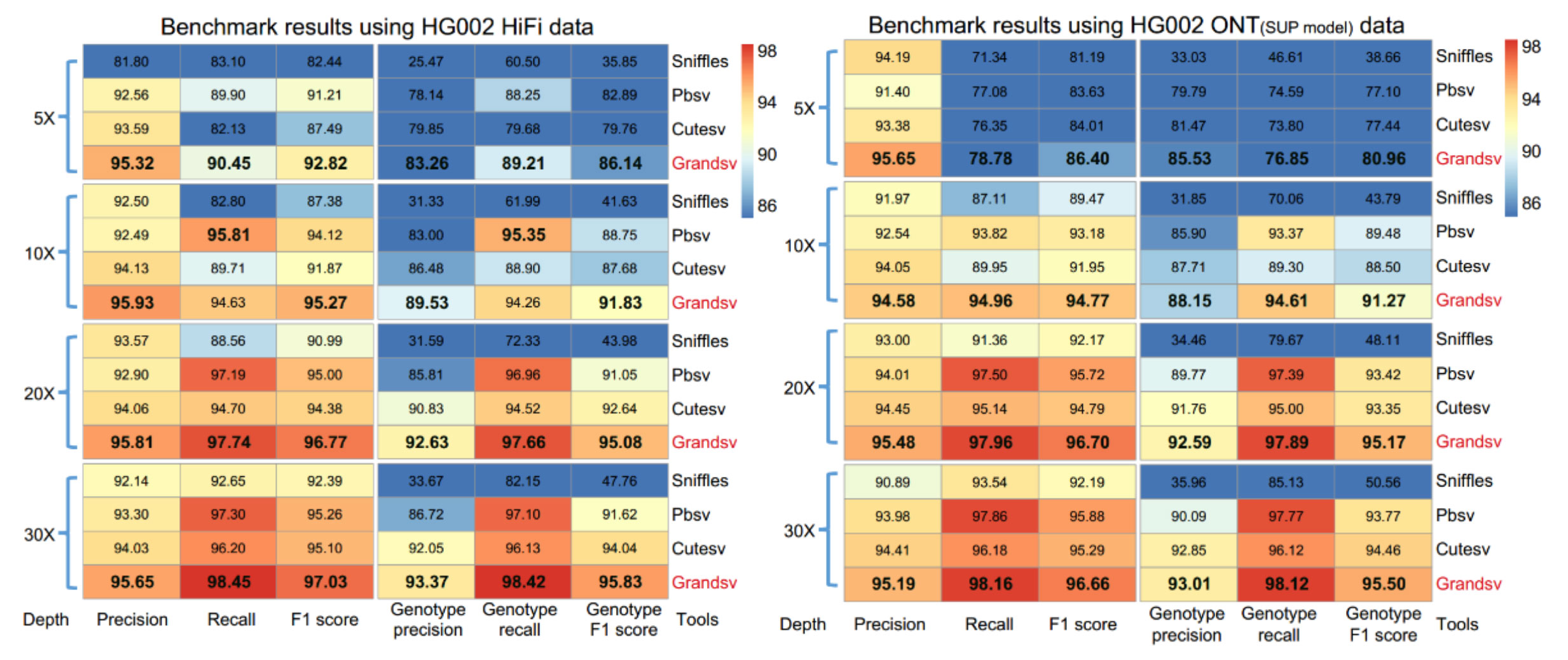
图2. Benchmark with HG002 PacBio HiFi and ONT data
相较于其他长读长SV检测软件,GrandSV有着更高的灵敏性。图3是HG002中一个长的片段插入,由于测序reads太短,单条read无法跨过,导致其他的软件在此处只能检出断点,而GrandSV通过局部组装可以完整的组装出跨过这个SV的一致性序列。图4是HG002中两个杂合的片段插入,其他软件只能检测出一个平均长度约94bp的片段插入,而GrandSV能够准确检测出两个不同的杂合的片段插入。
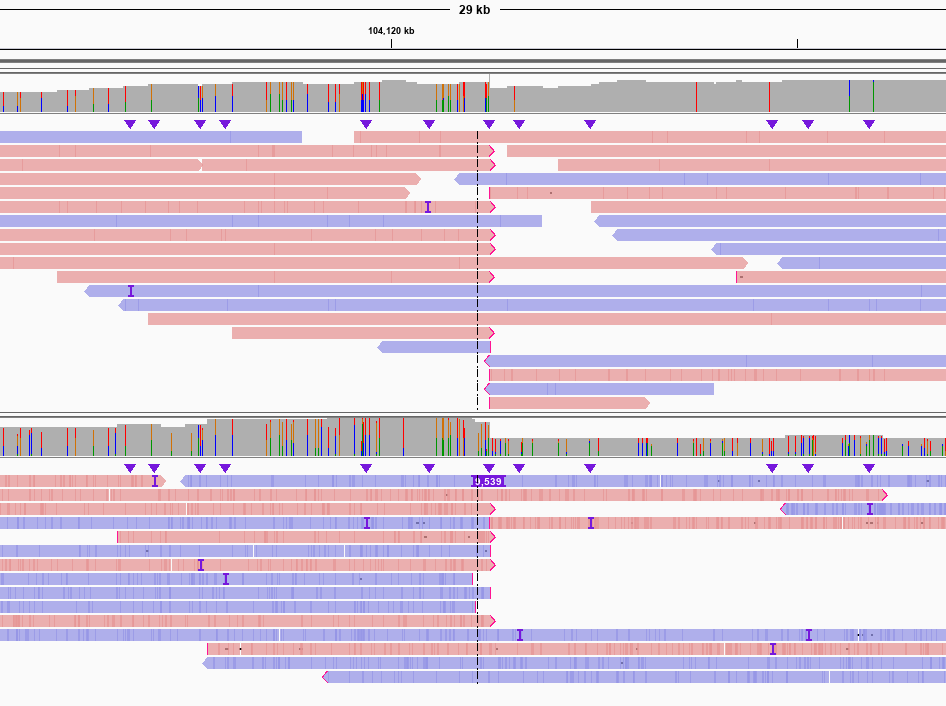
图3. Only GrandSV called correctly for a 9562 bp INS
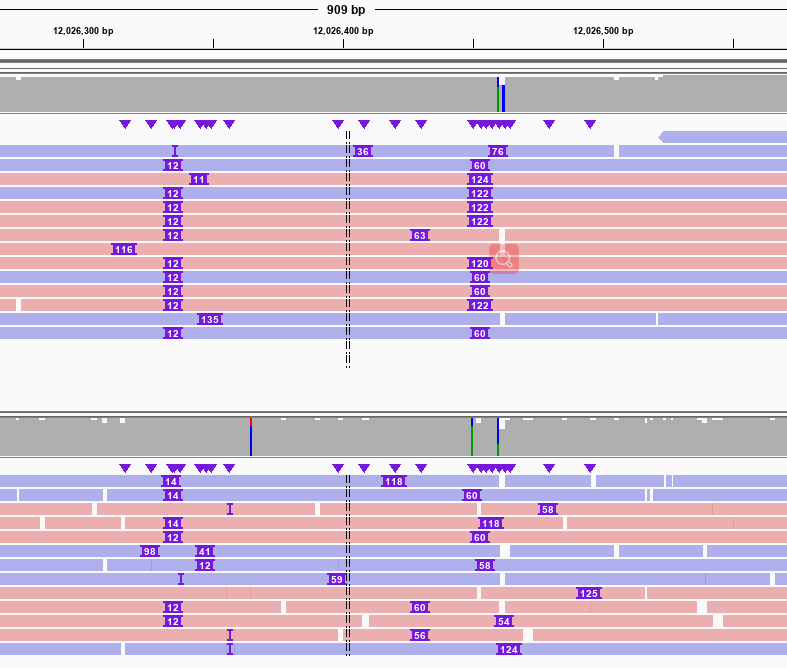
图4. Only GrandSV called correctly for a 60 bp INS and a 122 bp INS
Sentieon三代PacBio HiFi数据SNP/Indel检测软件——DNAscope LongReadsSentieon在二代测序中SNP/Indel变异检测流程已非常成熟,并以其检测准确性高和检测速度快而广受业内人士认可。近日,Sentieon推出了DNAscope LongReads分析流程,深度改进DNAscope流程,加入Sentieon分型(Phasing)模块,高速准确分析PacBio HiFi数据进行SNP/Indel检测。
DNAscope LongReads运算效率高,速度相比开源软件有很大的提升。其中比对模块Sentieon Minimap2与原版相比提速2倍,而变异检测模块与DeepVariant相比提速6倍,有助于用户提升交付速度,降低计算成本。
准确度方面,DNAscope LongReads流程获得了FDA挑战赛PacBio数据的两个分项冠军,SNP的F1 score达到了0.9993,Indel为0.9943。在低深度下对比10x PB HiFi,16x PB HiFi,30x Illumina的全基因组测试结果,可以发现全基因组范围内16x的HiFi数据的准确率就已经超越了30x Illumina的数据,在低复杂度的基因组区域内即使10x的HiFi数据也可以超越Illumina的准确度。
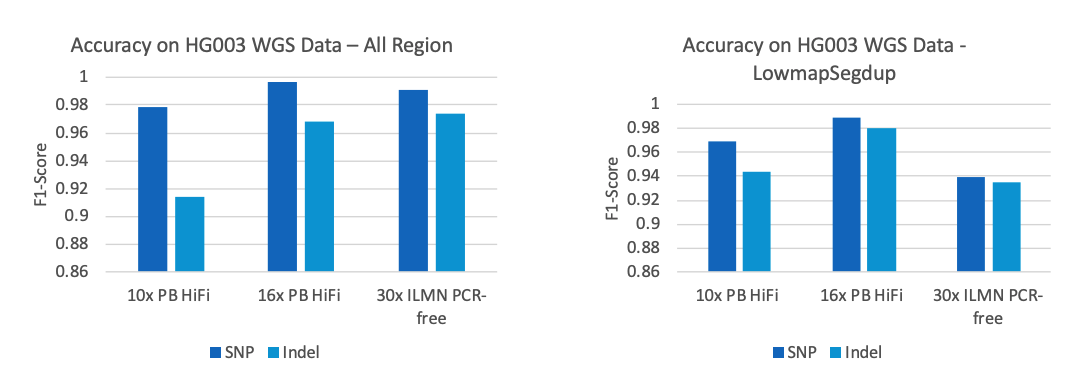
图5. SNP and Indel accuracy on HG003 WGS Data, PB HiFi vs ILMN
长读长数据可以覆盖Illumina序列所无法覆盖的区域,例如395个位于常染色体上复杂区域的临床相关基因(CMRG)。这些基因具有重要的临床价值,然而由于所处基因组区域较为特殊,短读长序列难以比对。以SMN1基因为例,该基因是脊髓性肌萎缩症的致病基因,最常见的突变是外显子7和8的缺失。从下图可以看出,只有PB HiFi数据可以覆盖相关区域,得出变异检测结果。
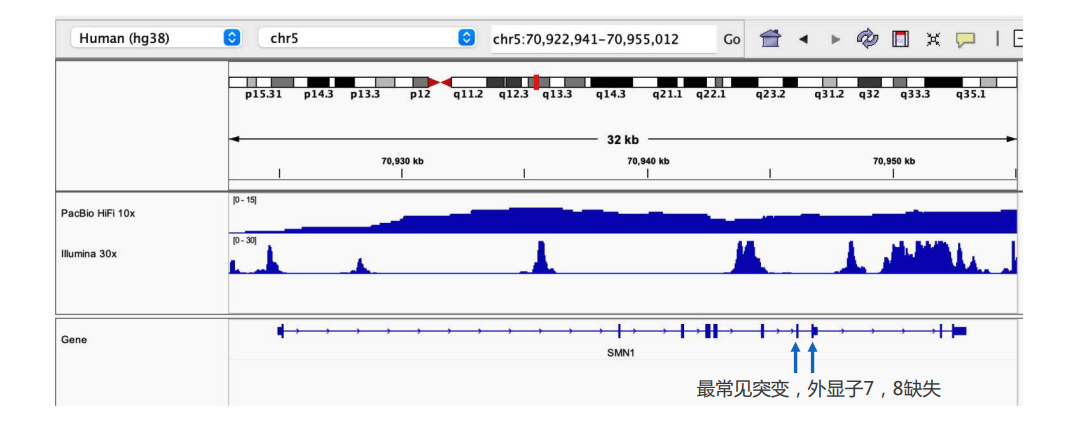
图6. PB HiFi covers SMN1 gene region when ILMN reads fail
通过结合双方在长读长变异检测流程中的特有优势,将点突变及大片段的结构变异整合分析,推出的完整HiFi全基因组重测序分析流程,将极大地加快PacBio HiFi测序的临床应用。基于本次成功经验,在未来,双方团队还会在Oxford Nanopore的数据处理上进行合作,为业界带来更多优质的Long Reads重测序的分析流程。同时双方也会持续在其他领域包括华大测序平台应用,以及“希望诊断计划”项目中保持合作,共同加速全基因组临床产品的市场教育和推广工作。







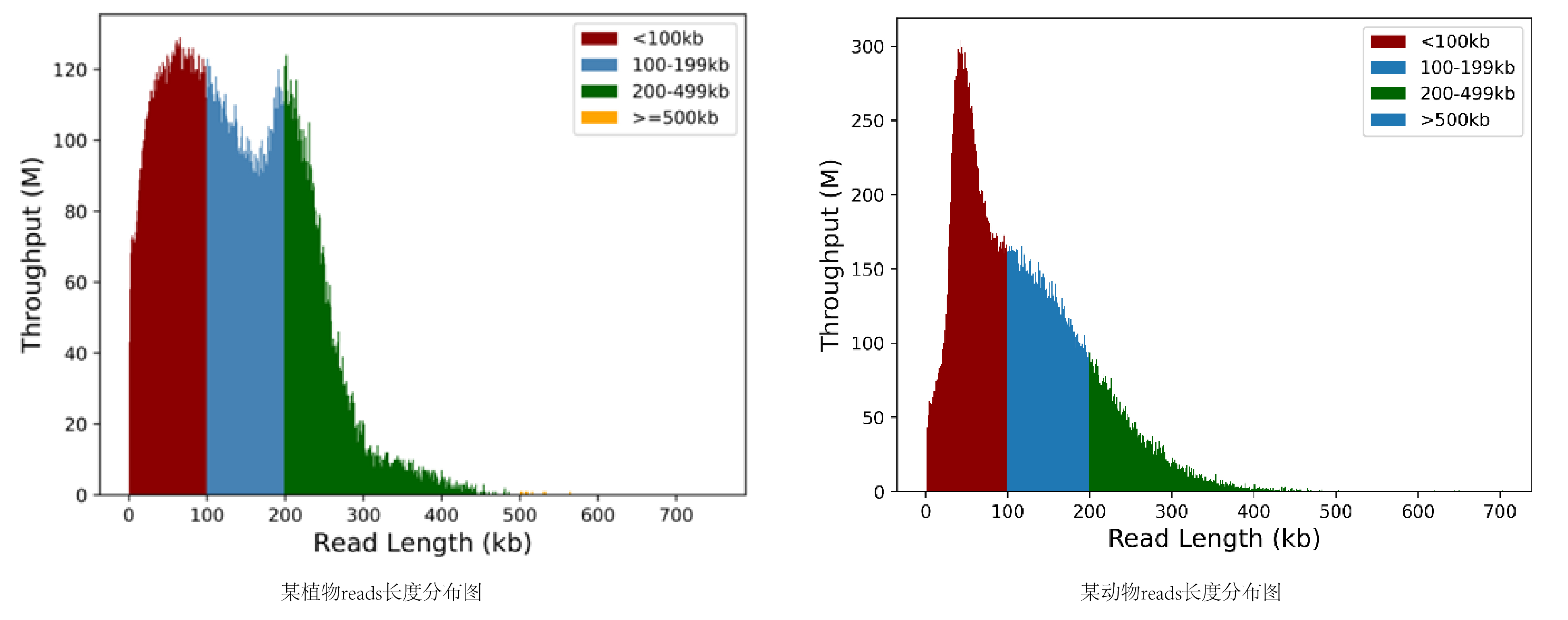
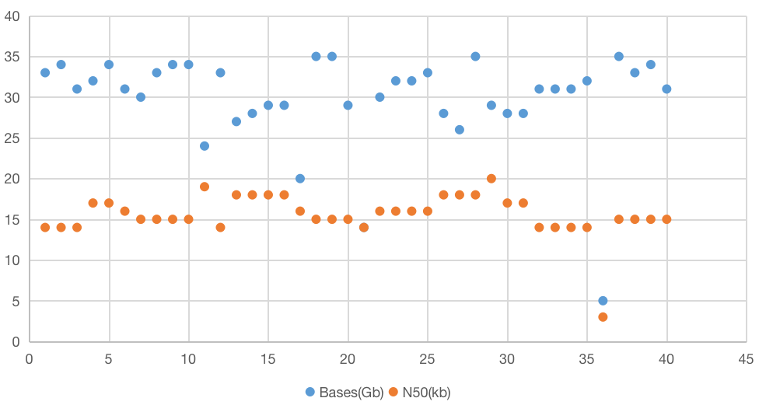 HiFi数据量和长度统计
HiFi数据量和长度统计















 希望组CEO汪德鹏(左二)现场领奖
希望组CEO汪德鹏(左二)现场领奖



